s |
focus search bar ( enter to select, ▲ / ▼ to change selection) |
g c |
go to cluster |
g e |
go to edctools |
g f |
go to facility |
g g |
go to guidelines |
g t |
go to training |
h |
toggle this help ( esc also exits) |

Journée de formation des enseignantes et enseignants de l’Académie de Créteil
Mercredi 7 décembre 2022
De 9h00 à 17h30

Introduction de l’unité
La directrice Valérie Mezger présentera l’unité. Retrouvez ici le support de sa présentation.
Ateliers
Vous allez participer à 3 ateliers qui ont pour but de vous donner un aperçu des méthodes utilisées au laboratoire pour étudier les génomes et leur régulation.
Ces ateliers sont animés par :
- Léo Carrillo
- Emmanuel Cazottes
- Laure Ferry
- Magali Hennion
- Olivier Kirsh
- Jean-François Ouimette
L’ADN : de l’extraction à la caractérisation
Dans cet atelier nous allons observer sur gel d’électrophorèse le produit d’amplification par PCR d’une région de l’ADN génomique issu de différents types cellulaires murins. Nous comparerons également les niveaux d’expression des transcrits provenant de cette même région dans les différents types cellulaires.
Les diapositives présentées sont disponibles ici.
L’analyse de la méthylation de l’ADN
Dans cet atelier nous allons mesurer le niveau global de méthylation de l’ADN génomique issu de différents types cellulaires murins. Pour cela nous utiliserons la technique LUMA (LUminometric Methylation Assay), basée sur le Pyroséquençage des produits de digestion par des enzymes sensibles à la méthylation.
Les diapositives présentées sont disponibles ici.
La bio-informatique
Avec l’essor du séquençage à haut-débit, les besoins en analyses de données ont explosés dans les laboratoires de biologie. En tout premier lieu, il y a un besoin de visualisation des données qui sortent du séquenceur sous la forme de fichiers texte de plusieurs dizaines voire centaines de millions de lignes. Dans cet atelier, nous allons utiliser un logiciel pour visualiser les données de séquençage après alignement sur le génome.
Les diapositives d’introduction présentées sont disponibles ici.
Les données de test (700 MB) peuvent être téléchargées ici si vous voulez réessayer chez vous. Un fichier igv_session.xml est inclus dans ce dossier et permet de charger directement toutes les données dans IGV (Menu File/Open Session…).
Le logiciel IGV est disponible ici.
Navigateur de génome
- Ouvrir IGV
- Choisir le génome : souris (mouse), version mm10
- Cliquer sur un chromosome, observer la piste “Refseq genes”, zoomer en cliquant sur le “+”. Cette piste indique la position des gènes.
- Taper “actb” dans la barre puis cliquer sur “Go”.
- Avec un clic droit sur la piste Refseq, il est possible de changer le format d’affichage (Collapsed, Expanded, Squished).
Observation de données d’expression
- Ajouter les données de transcription: Menu File/Load from File… Dossier “expression”.
- ESC_SRR14102760.bw
- MEFs_SRR14102762.bw
- Pour que ce soit plus lisible:
- changer les couleurs (ESC = bleu, MEFs = vert): clic droit sur le nom de la piste, puis “Change Track Color…”
- changer la hauteur des pistes: Menu Tracks/Set Tracks Height… -> 80 (ou Fit Data to Window)
- pour que les données soient à la même échelle: sélectionner les 2 pistes (Ctrl+clic), clic droit, “Group Autoscale”.
- Se ballader, zoomer, aller voir les gènes:
- Rfx2
- Rps24
- Sox2
- Fst
- Vim
Observation de données de méthylation de l’ADN
- Ajouter les données de méthylation: Menu File/Load from File… Dossier “methylation”.
- ESC_me_GSM6070571.bw (entre 0 et 1)
- MEFs_me_SRR8146641.bw (entre 0 et 100)
-
Ajuster les échelles (clic droit, “Set Data Range …”) et changer la couleur de la piste MEFs.
- Ajuster l’affichage : Menu Tracks/Fit Data to Window
- Ajouter la piste de la densité en dinucléotides CG (CpG_density.bw) et celle des îlots CpG (CpG_Islands.bed). Changer les couleurs selon vos goûts.
Votre fenêtre devrait maintenant ressembler à ça:
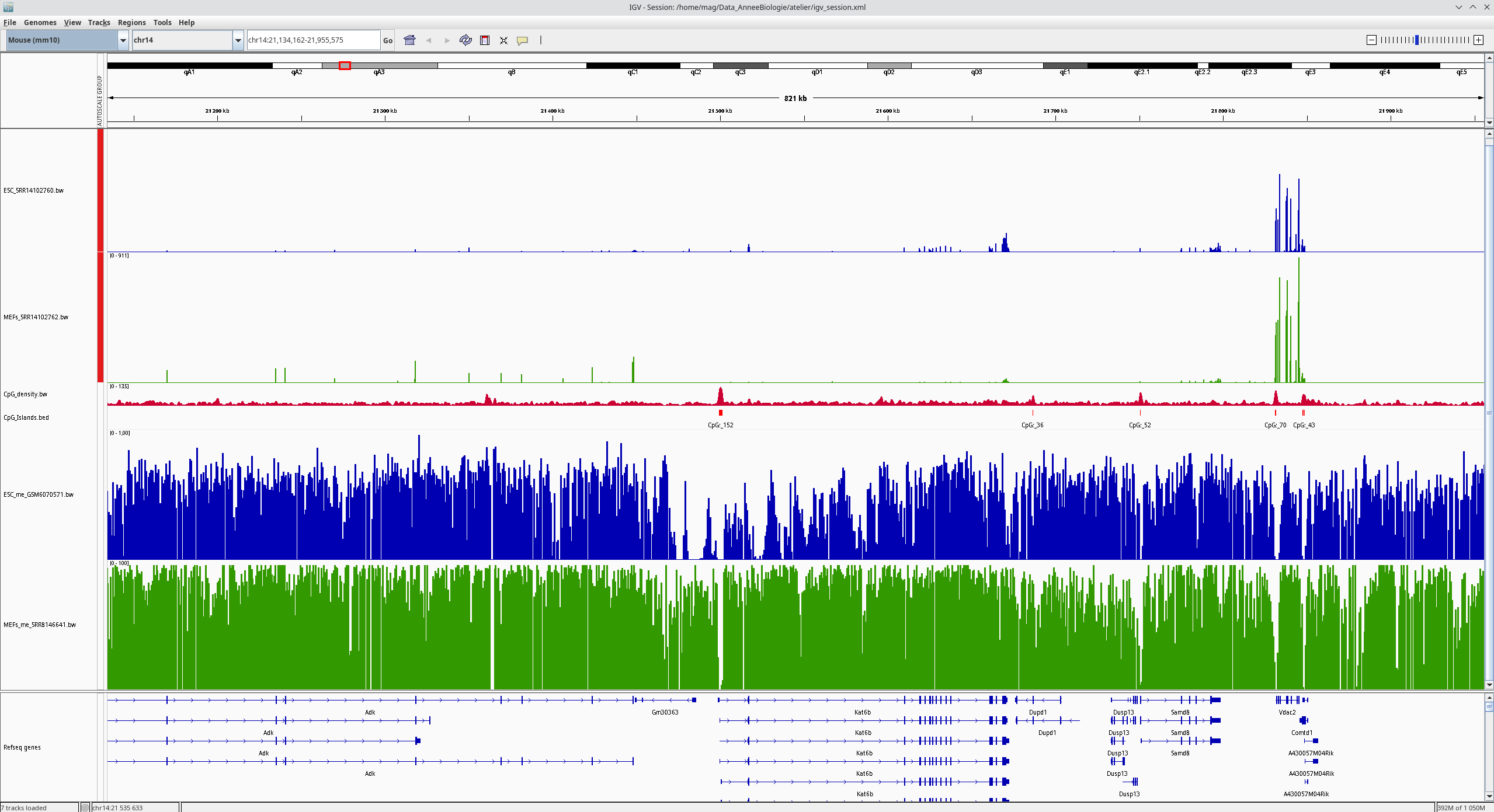
- Aller voir quelques gènes comme:
- Acta2
- Sox2
- Ezh1
- Vim
- Rfx2
- Dazl
Observer l’expression et la méthylation (au niveau des promoteurs).
Information sur les gènes observés
- Acta2 actine alpha
- Atf4 Activating transcription factor 4, facteur de transcription
- Ezh1 composant du complexe Polycomb PRC2 (méthylation de la lysine 27 de l’histone H3)
- Fst Follistatine, régulation des facteurs de croissance de la famille de TGFβ
- Rfx2 regulatory factor X 2, regulation de la transcription par l’ARN polymérase II
- Rps24 protéine ribosomique (40S)
- Sox2 facteur de transcription essentiel dans le maintien de l’auto renouvellement des cellules souches
- Vim vimentine, filaments intermédiaires faisant partie du cytosquelette
Vous pouvez trouver plus d’information sur les gènes sur les bases de données en ligne, comme celle de Ensembl.
|
BiBs
2025 parisepigenetics
https://github.com/parisepigenetics/bibs |
| programming pages theme v0.5.22 (https://github.com/pixeldroid/programming-pages) |